Podría identificar, en una sola prueba, COVID, influenza y virus sincicial respiratorio; incluso dengue, chikungunya y Zika
Ciudad de México.- De manera más rápida y precisa será posible detectar y cuantificar el virus del SARS-CoV-2, causante de la pandemia de la COVID-19, con una metodología desarrollada por un grupo interdisciplinario de investigación de la UNAM, del Instituto Politécnico Nacional (IPN) y del Instituto Mexicano del Seguro Social (IMSS).
Primero en muestras de estándares sintéticos y después en muestras de pacientes, mediante un dispositivo microfluídico que en un futuro facilitará realizar pruebas fuera de los laboratorios, es decir, en clínicas y centros de salud con atención médica básica.
La metodología fue publicada en un artículo científico de la revista Microsystems & Nanoengineering, perteneciente al grupo editorial Nature.
El proyecto -donde se conjuntan las capacidades de infraestructura de cada institución para abordar diversos aspectos- lo integraron:
Luis Fernando Olguín Contreras, académico de la Facultad de Química (FQ); Laura Oropeza Ramos, docente de la Facultad de Ingeniería (FI); Luis Álvarez Icaza y Óscar Pilloni Choreño, investigadores del Instituto de Ingeniería (IIngen); Kenia Chávez Ramos, Frida Trejo y Prisciluis Caheri Salas Navarrete, posdoctorantes en la Universidad Nacional. Asimismo, Eva Ramón Gallegos, del IPN; y José Esteban Muñoz Medina, del IMSS.
Más aplicaciones
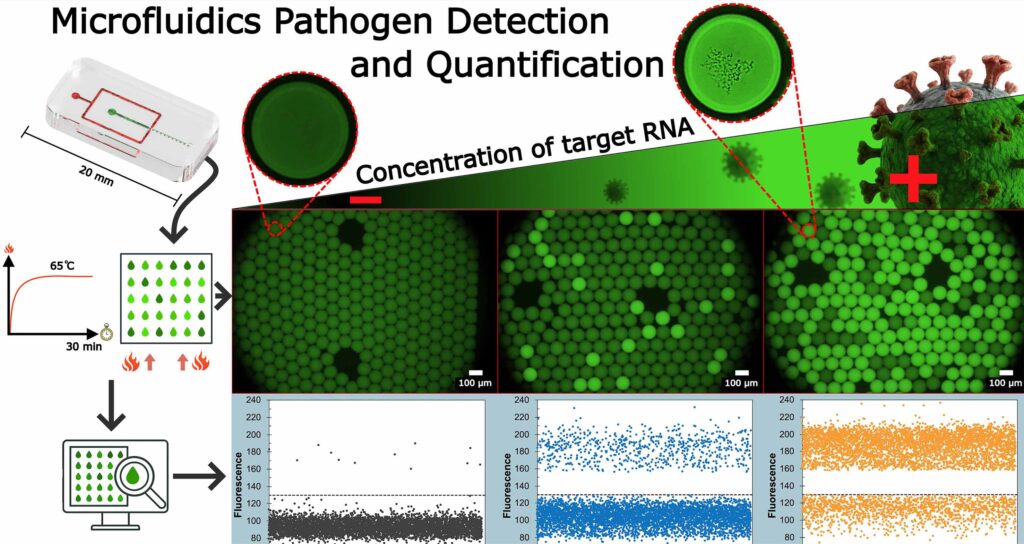
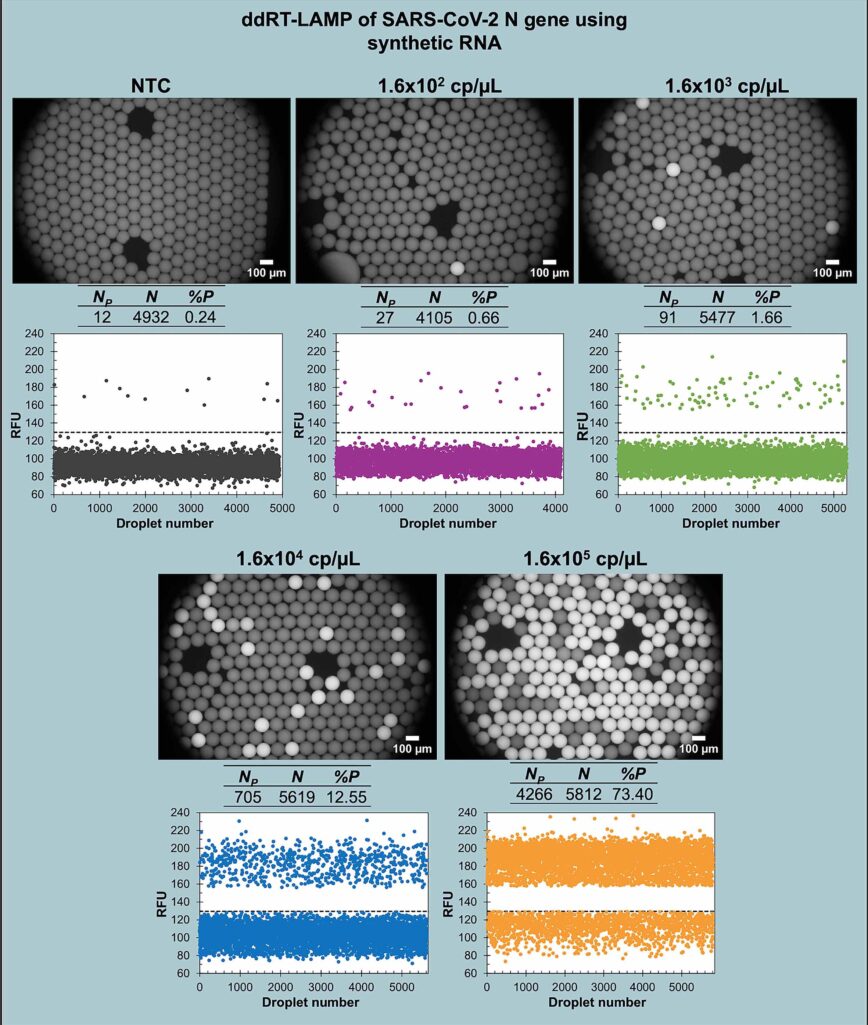
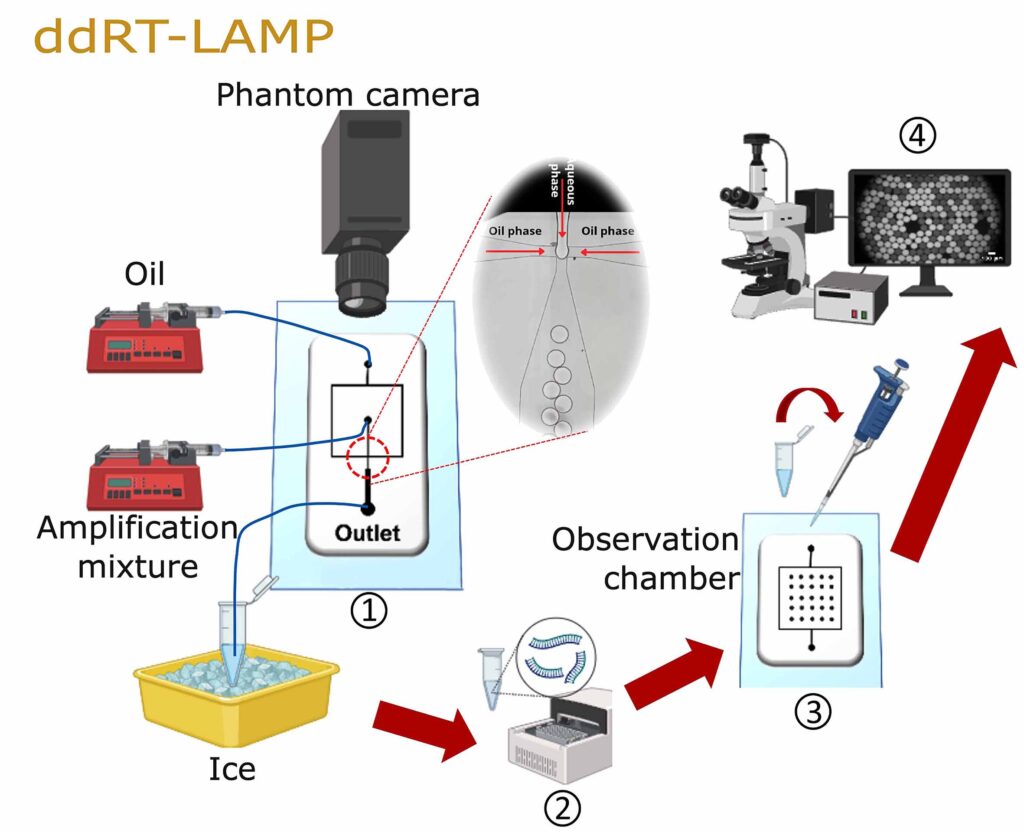
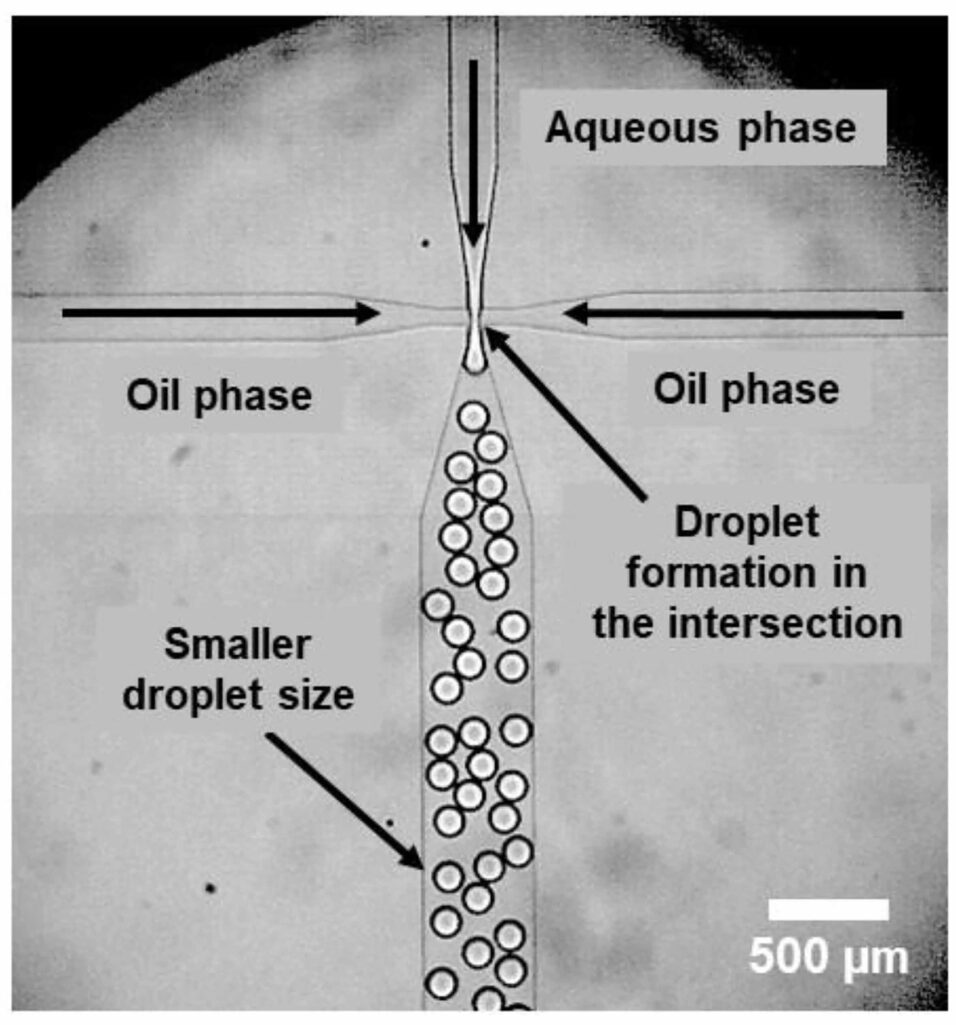
La microtecnología propuesta permite dividir una muestra del paciente en miles de microgotas utilizando un dispositivo microfluídico. En cada una se realiza una reacción bioquímica y después se analiza el porcentaje de las positivas para determinar de forma absoluta el número de partículas virales, explicó Kenia Chavez.
Además, la reacción bioquímica utilizada en la detección se produce a una temperatura constante. En cambio, la PCR, que es el patrón de referencia, requiere cambios de temperatura drásticos. Con ello podemos llevar a cabo la detección del SARS-CoV-2 en un ensayo con una duración de 30 minutos, en vez de las dos horas empleadas en la PCR, acotó Luis Olguín.
Es el primer paso que se ve plasmado en una publicación importante a nivel internacional, indicaron Laura Oropeza y Luis Álvarez Icaza.
En México, destacaron Caheri Salas y Esteban Muñoz, “circulan tres virus respiratorios de importancia: COVID, influenza y virus sincicial respiratorio; entonces, este trabajo podría ser modificado para detectarlos en una sola prueba, lo cual representaría un ahorro económico relevante. Otras enfermedades que sería capaz de localizar son el dengue, chikungunya y Zika.
Oscar Pilloni resaltó que una de las aplicaciones será contar con un equipo portátil, “factible de llevar a lugares en donde no se cuenten con laboratorios de biología molecular para diagnóstico, así como realizar pruebas para mayor cantidad de enfermedades y, en un solo ensayo, tener el diagnóstico para varios padecimientos”.
A decir de Laura Oropeza, subraya la importancia de la interdisciplina donde todas las áreas son fundamentales, en este caso se laboró a partir de la ingeniería, microfluídica, microfabricación, bioquímica, biotecnología y la medicina, entre otras. Además, la publicación es fundamental porque amplía la posibilidad de llevarlo a un proceso tecnológico.
En el artículo titulado Critical aspects of droplet digital reverse transcription loop-mediated isothermal amplification (ddRT-LAMP) for viral pathogens detection (Aspectos críticos para la detección de patógenos virales utilizando la transcripción reversa y la amplificación isotérmica por bucle digital en microgotas [Chavez et al Microsyst Nanoeng 11, 137, 2025]) participaron, por parte de la Universidad Nacional las investigadoras e investigadores de la FQ y de FI, así como del IIngen. (UNAM)